脳腫瘍病理
脳腫瘍連携研究
脳腫瘍のゲノム・エピゲノム統合診断とは
はじめに
がんは、紀元前から認識されており、「すべての細胞は細胞から(omnis cellula e cellula)」1という箴言を唱えたドイツの病理学者Rudolf Virchow(1821–1902)の時代から、がん細胞の形態学的観察がなされてきました。同時期にがんの外科治療や放射線治療も試みられ、治療戦略が多様化したため、がんの治療効果の最大化と予後予測の要請により、1900年代に入ってから形態学的病理診断、即ち外科病理学が体系化されてきたものと考えらます2。今でも形態学的診断が病理診断の基本であることに変わりはありませんが、がん種類や診断目的によっては電子顕微鏡による超微細構造の観察、免疫組織染色によるタンパク発現とその局在、そして分子遺伝学的解析によるバリアント解析やゲノム構造異常などの検索も加わり、現代では病理診断とは、「種々の診断技法を駆使した総合的な判断」としての医行為と言えます。ここでは近年、病理診断体系が形態診断からゲノム・エピゲノム統合診断に移行した脳腫瘍の病理診断について、当教室での取り組みとともに紹介します。
脳腫瘍診断のパラダイムシフト
頭蓋骨や血液脳関門といった解剖学的制約や、その低い発生頻度により、治療に難渋する症例が多く、脳腫瘍は最も予後不良な腫瘍の一つです。新規治療開発においては、治療対象を特定し、その本質を捉えるために、頑健にして適切な分類体系を必要としており、脳腫瘍分類は1926年にBaileyとCushingが神経上皮の発生過程との形態学的類似性にもとづき、その原型を作り3、2007年に出版された世界保健機構 (World Health Organization: WHO)が発刊するWHO分類第4版まで、形態学的な特徴に基づいた分類が採用されてきました4。より厳格で客観的な腫瘍の定義を目指し、WHO分類改訂第4版(2016年)では遺伝子―形態統合診断が導入され5 、さらにWHO分類第5版ではDNAメチル化分類も統合されました6。現在、脳腫瘍診断は、形態ーゲノムーエピゲノム(DNAメチル化)のマルチモーダルな分類体系に基づく、分子―形態統合診断となっています。
分子―形態統合診断は、形態学的所見に加え、腫瘍を特徴づける遺伝子異常や染色体異常を考慮するアプローチであり、より厳格に、再現性高く分類することが可能です7。例えば、最も頻度の高い原発性脳腫瘍である成人型浸潤性膠腫は、IDH(IDH1, IDH2)変異と1番染色体短腕と19番染色体長腕のヘテロ接合性欠失(1p/19q共欠失)の有無に基づき、Astrocytoma, IDH-mutant(星細胞腫, IDH変異)、Oligodendroglioma, IDH-mutant and 1p/19q-codeleted(乏突起膠腫, IDH変異および1p/19q共欠失)、およびGlioblastoma, IDH-wildtype(膠芽腫, IDH野生型)の3つに分類されます(下図 cBioPortalより改変)。
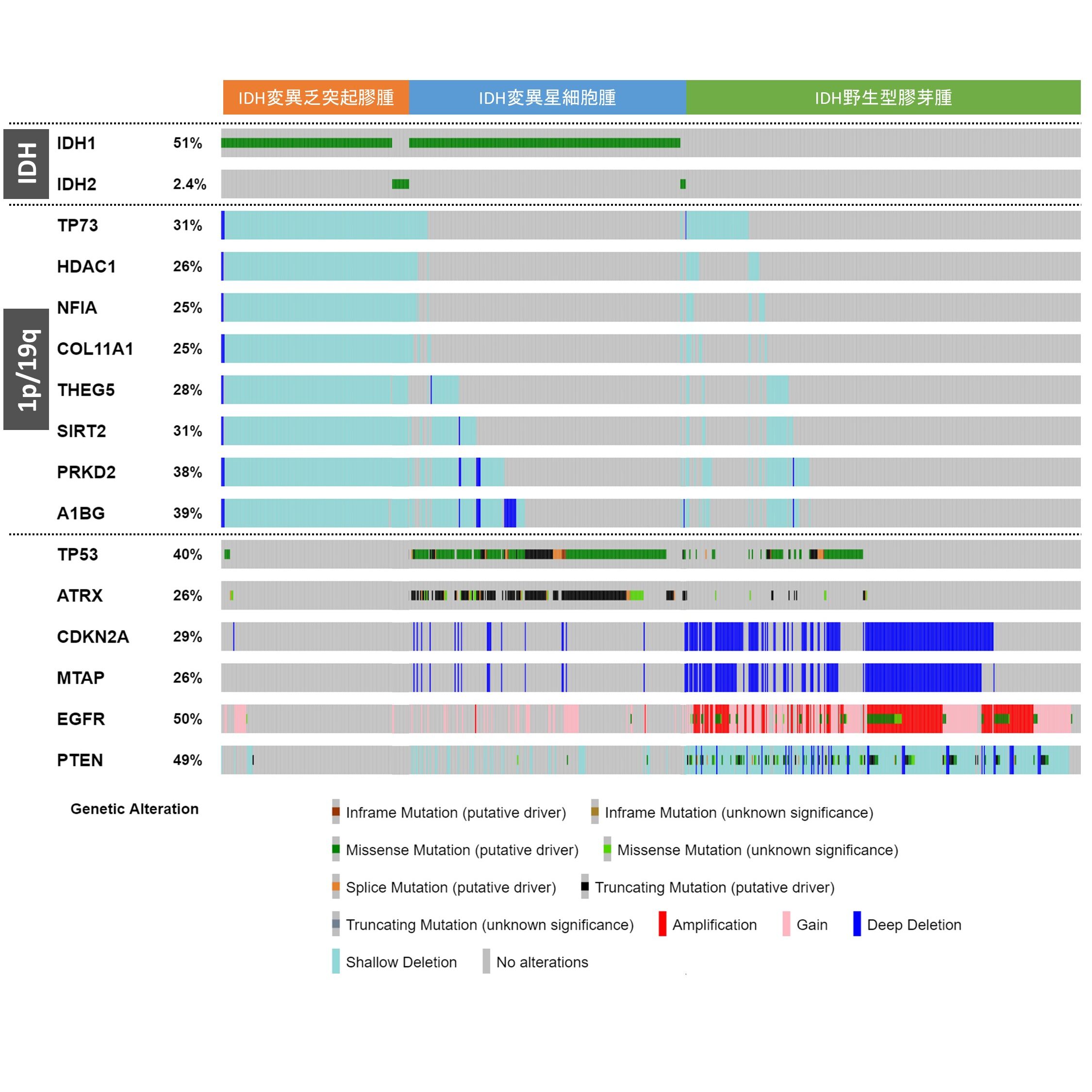
これにより、星細胞腫は核の大小不同、核形不整が目立ち、核クロマチンが濃染する一方、乏突起膠腫は核クロマチンが粗造となった類円型核を有し、核周囲ハローがみられるといった形態学的な特徴だけでなく、分子遺伝学的特徴を加味した統合診断が行われます。なお、膠芽腫, IDH野生型は、従来、形態学的所見(微小血管増殖、壊死)によって規定されてきたが、分子所見(TERTプロモーター変異、EGFR増幅、染色体7番gain/10番loss [7番トリソミー/10番モノソミー])も定義に加わり、二つの形態学的定義を満たさない場合でも、三つのうちいずれかの分子所見があれば膠芽腫, IDH野生型と診断が可能となったことで、分子遺伝学的定義により腫瘍概念が拡張されたといえます(下図、文献7より改変)。
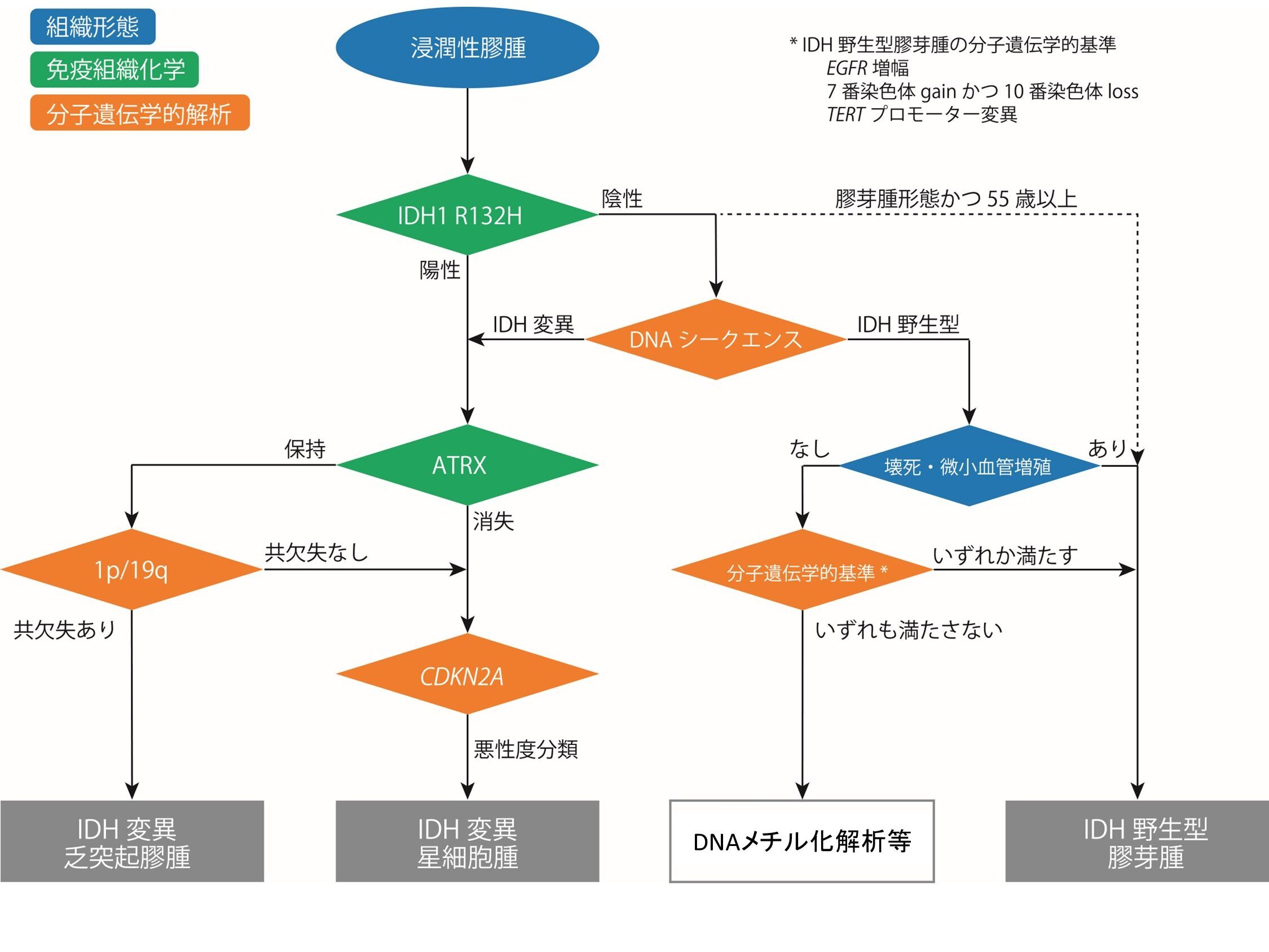
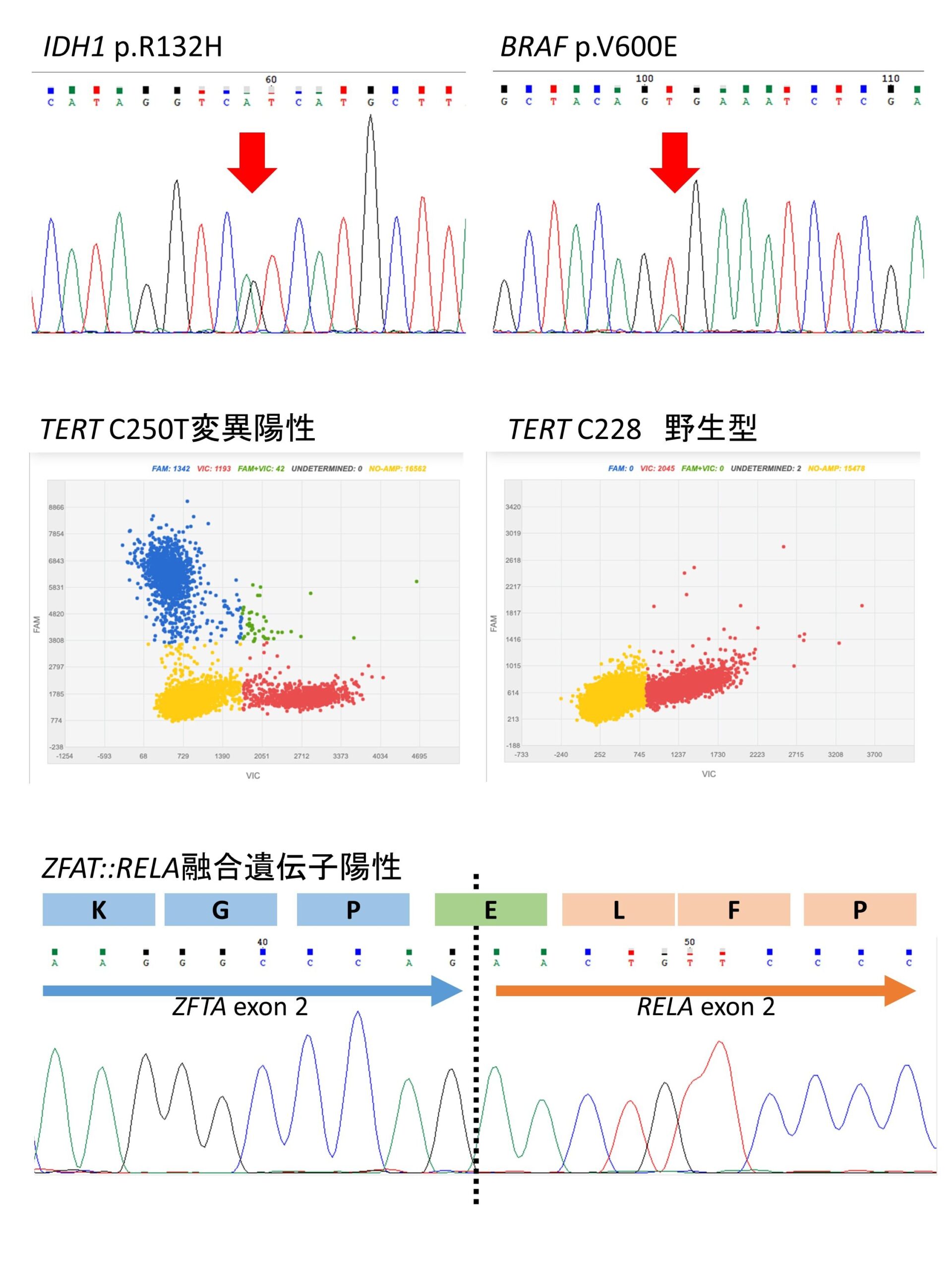
WHO分類に収載されているだけでも、脳腫瘍の腫瘍型は200を越えます。浸潤性膠腫をはじめ、その多くは統合診断にあたり分子遺伝学的検索が要求されるものの、日常の病理診断のなかで、全ての検索項目に逐一対応するのは現実的ではありません。脳腫瘍の統合診断では、免疫組織化学やFISH法による分子遺伝学的検索の代理が試みられているほか、当教室では形態学的検索を優先し、真に必要とされる項目に絞って分子遺伝学的検索を行なっています。具体的には、浸潤性膠腫ではIDH1, IDH2, H3-3A, H3C2, BRAF, TERTの各遺伝子の変異解析を、サンガーシークエンス法およびデジタルPCR法 (QuantStudio® 3D Digital PCR System TaqMan® Liquid Biopsy dPCR Assays, Thermo Fisher Scientific)によって検索しています。また、その他の一部の脳腫瘍では、形態学的診断を補助する目的で、Reverse Transcription (RT)-PCR法による融合遺伝子検索も行なっています。さらに、近年、注目されているDNAメチル化分類も必要に応じて積極的に検索しています8(下図、文献8より改変)。
DNAメチル化分類
脳腫瘍分類の世界標準であるWHO分類第5版から新たに導入されたDNAメチル化分類9は、統合診断精度の改善に寄与し、BaileyとCushing以来のもっとも重要な改革と評されています10 。DNAメチル化は、シトシンとグアニンが並ぶCpGのシトシン残基の5位の炭素原子にメチル基修飾が付加される化学反応であり、種々の機序により遺伝子発現調節を介して生命現象に関与します。ゲノムワイドなDNAメチル化状態、すなわちDNAメチロームは個体発生において細胞分化を制御するほか、腫瘍発生においても細胞起源を反映したプロファイルを呈することを利用して、Capperらは、ゲノムワイドDNAメチル化アレイ[Infinium HumanMethylation450 BeadChip (450k) array (Illumina, San Diego, USA), Infinium HumanMethylationEPIC BeadChip (EPIC) array]を用いた、脳腫瘍のDNAメチル化分類を確立しました。非線形次元削減法であるt-distributed stochastic neighbor embedding (tSNE)によって、脳腫瘍は独立したクラスタを形成することが明らかとなり、さらにその多くは統合診断によって確立されてきた腫瘍型にほぼ合致することを示し、DNAメチロームをランダムフォレスト法によって10,000の分岐を経てDNAメチル化分類を推定可能な機械学習法、いわゆるAIによって作成し、ドイツがんセンター(Deutsches Krebsforschungszentrum, DKFZ)のウェブサイトによってウェブツールとして公開しています (DKFZ classifier, https://www.molecularneuropathology.org/mnp/) (文献9より改変)。
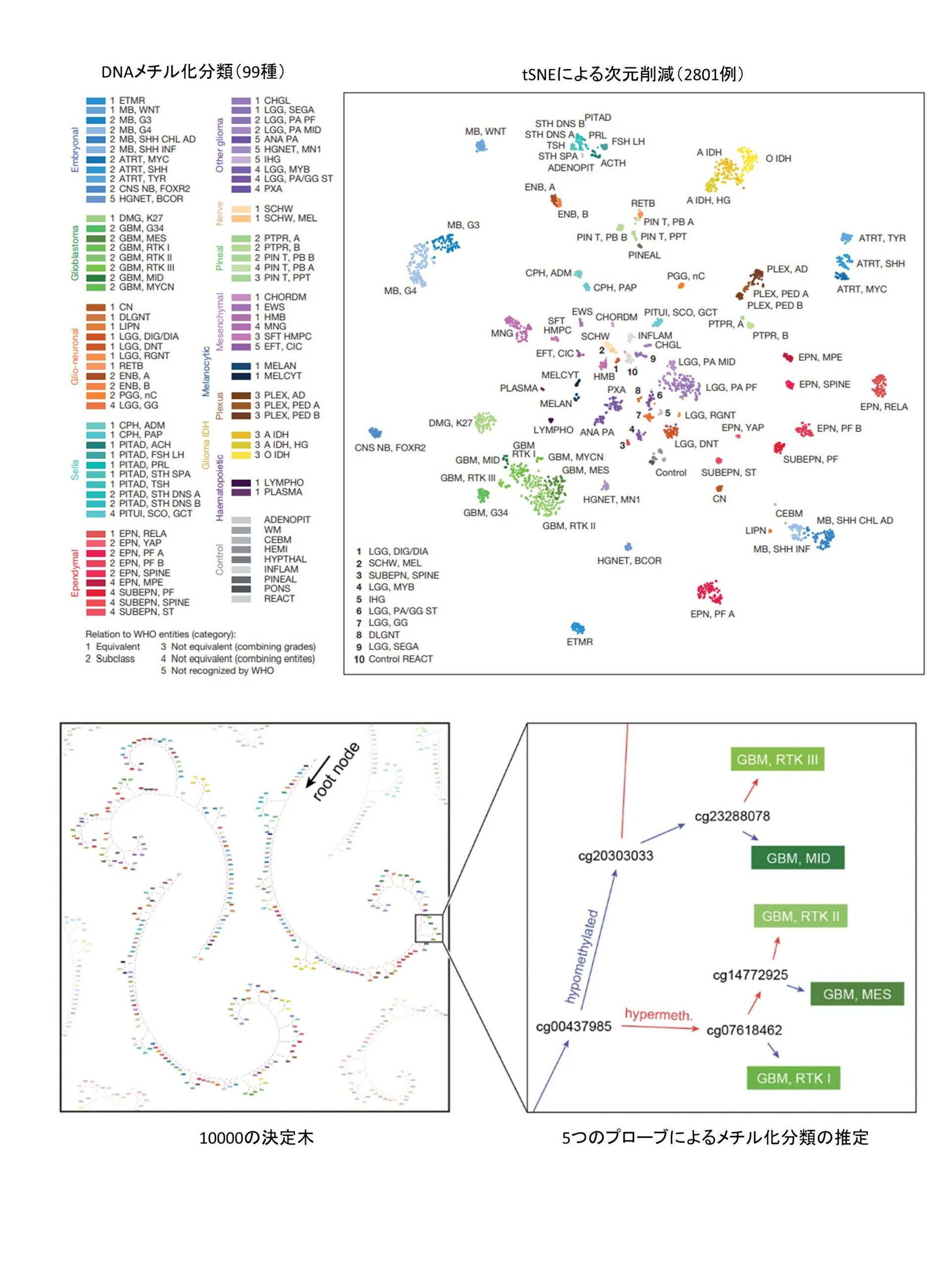
その原理であるtSNEについて、詳しく解説します。DNAメチル化アレイによって得られる脳腫瘍のDNAメチロームは、プローブ数(85万ないし95万)の次元数を有する高次元データであるため、そのままではデータ特性を人間の認知能では把握できません。そこで、適切な次元削減法による可視化が必要となります。従来用いられてきた次元削減法のうち、線形次元削減法であるPrincipal component analysis (PCA)11は、脳腫瘍のDNAメチロームを次元削減してもぼんやりとクラスタしか見いだせない一方、非線形次元削減法であるtSNE12や、Uniform Manifold Approximation and Projection (UMAP)13であれば、明確なクラスタが形成されます(下図、文献8より改変)。
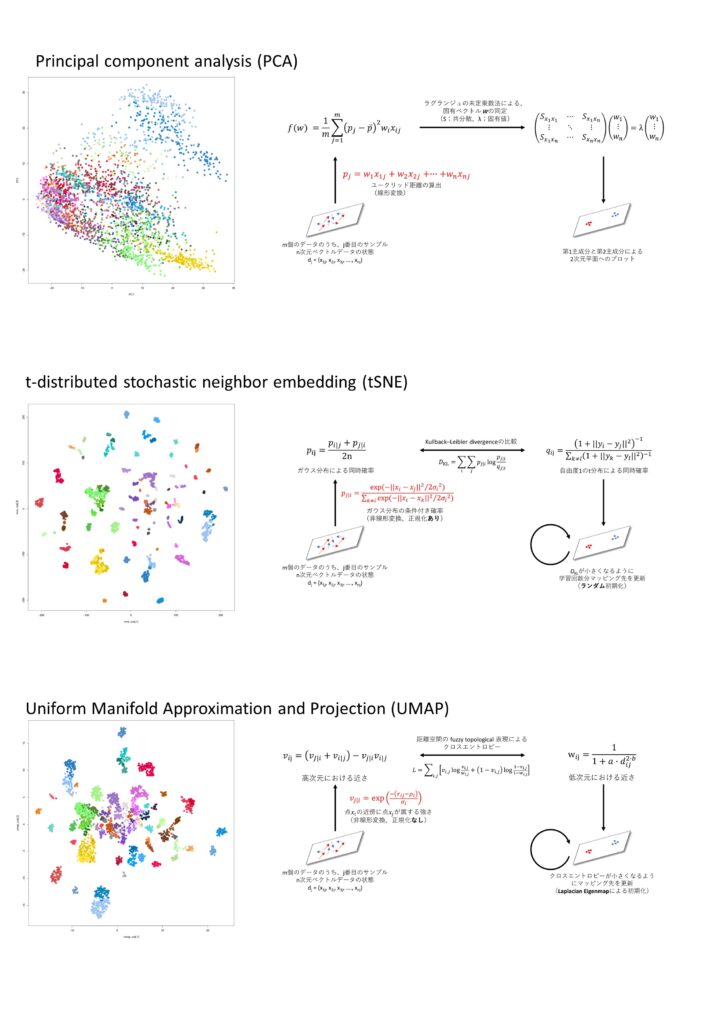
tSNEは、サンプル同士の類似度(Local structure)を優先して2次元平面(ないし3次元空間)にプロットするため、低次元空間では直感的なユークリッド距離による比較は困難で、プロットの集合である各クラスタを、どこまでを同一クラスタとみなすか断定は困難であるほか、クラスタ同士の距離をもってクラスタ間の類似性(Global structure)を議論することも困難ですが、脳腫瘍のDNAメチロームの特性の可視化には重要な役割を果たします。当教室では、脳腫瘍14だけでなく軟部肉腫においてもDKFZ classifierの実臨床上の限界と、tSNEが統合診断に寄与することを示しており15、DKFZ classifierだけに頼らないDNAメチル化解析に取り組んでいます。
なお、UMAPは、埋め込み次元数によらず実行時間がほぼ一定で、既存の学習空間に新規サンプルを埋め込み可能であることに加え、Global structureが保たれやすいとされています16。UMAPは、観測されるデータがリーマン多様体の上に一様分布すると仮定し、それをfuzzy topological表現により、我々が認知可能な低次元ユークリッド空間にマッピングする手法です。Fuzzy topological表現は、原著論文においては圏論 (Category Theory) の手法で説明されています。腫瘍分類とは、class discovery(疾患の新たなカテゴリーを正確に認識する過程)とclass prediction(個々の症例が既知のどのカテゴリーに属するかを決める過程)いう明瞭に異なる二つのプロセスが包含されており、病理学は最初から疾患概念そのものを標的とし、未知の疾患をも標的とする学問分野であるとされ17、対象とその間の射から構成される「圏」を考察の対象とする圏論が、対象の類似性や関係性を抽象的に捉えるための方法だとすれば18、UMAPによる脳腫瘍のDNAメチロームの可視化は、脳腫瘍における未知の生物学的本質をつくようで興味深く思っています。
さらに、DNAメチローム解析から、ゲノムワイドなコピー数解析が可能です。例えば、1p/19q共欠失, CDKN2A/Bホモ接合性欠失, EGFR増幅, 染色体7番gain/10番loss等は、統合診断における有用な支援ツールとなる。さらに、KIAA1549::BRAFやFGFR3::TACC3のような、重複領域を伴う融合遺伝子の推定も可能です。DNAメチロームから算出されるコピー数は、正常対照サンプルとの相対比であり、SNPアレイ等とは異なり、両アリルの平均値である。Rを用いた、コピー数を算出するためのパッケージは複数公開されており、特にconumeeはDKFZ classifierの報告書におけるコピー数プロットでも用いられるパッケージです。EPICアレイのアノテーションファイルに記述されている、ゲノム上のプローブ位置の情報と突合させれば、染色体腕ごと19、遺伝子ごと20のコピー数の平均も算出可能であり、研究手法としても有用です。
脳腫瘍診断のこれから
本邦では、病理診断のための分子遺伝学的解析であっても保険収載されておらず、そのほとんどは研究室レベルでの解析で対応しているのが現状です。しかしこれらの検索はすべての施設で行えるわけではなく、全国均てん化は困難であり、医療の先進性と現実性のバランスを取る必要があります。DNAメチル化分類は脳腫瘍の統合診断に大きな力を発揮し、本邦でも事実上の標準技術として認知されつつありますが、技術的、費用的制約から容易に行える解析ではなく、その病理診断における取り扱いも厳密には定まっていません21。現状では分子遺伝学的解析の品質や解釈は、各病理医が全責任を負い、統合診断として病理診断報告書に記載しています。WHO分類に厳格に従おうとすれば、分子遺伝学的検索なしに確定診断ができなくなった現在、最低限の分子遺伝学的解析は臨床検査として保険診療下に行われるべきと考えます。
脳腫瘍分類は今後もさらにゲノム・エピゲノム情報を統合して発展していくことは確実で、精細な脳腫瘍分類の追求は絶対的に必要不可欠な診療上の要請です。当教室では、これからも脳腫瘍診療の発展に寄与できるよう活動を続けてまいります。
参考文献
- Virchow. 細胞病理学. (川梶昭翻訳). 東京: 朝日出版社; 1988. ↩︎
- シッダールタ・ムカジー. がん─4000年の歴史─. (田中文翻訳): 早川書房; 2016. ↩︎
- Bailey P Cushing H. A Classification of Tumors of the Glioma Group on a Histogenetic Basis with a Correlated Study of Prognosis. Philadelphia: JB Lippincott; 1926. ↩︎
- WHO Classification of Tumours of the Central Nervous System. Louis DN OH, Wiestler OD, Cavenee WK, editor. Lyon: International Agency for Research on Cancer; 2007. ↩︎
- WHO Classification of Tumours of the Central Nervous System. Louis DN OH, Wiestler OD, Cavenee WK, editor. Lyon: IARC; 2016. ↩︎
- Louis DN, Perry A, Wesseling P, Brat DJ, Cree IA, Figarella-Branger D, et al. The 2021 WHO Classification of Tumors of the Central Nervous System: a summary. Neuro Oncol. 2021;23(8):1231-51. ↩︎
- 里見介史、市村幸一. 脳腫瘍. 病理と臨床. 【がんゲノム医療時代の分子腫瘍学】(第3部)がんの分子病理学(各論) 臓器がん 40. 東京: 文光堂; 2022. p. 168-77. ↩︎
- 里見介史、柴原純二. がん病理診断の潮流 形態診断からゲノム・エピゲノム統合診断へ. 杏林医学会雑誌 2023;54(4):227-234. ↩︎
- Capper D et al. Nature. 2018;555(7697):469-74. ↩︎
- Paulus W. Acta Neuropathol. 2018;136(2):177-9. ↩︎
- Stewart S et al. Chem Soc Rev. 2014;43(1):70-84. ↩︎
- LaurensvanderMaaten G. VisualizingDatausingt-SNE. Journal of Machine Learning Research. 2008;9:2579-605. ↩︎
- Leland McInnes JH, James Melville. UMAP: Uniform Manifold Approximation and Projection for Dimension Reduction. ArXiv e-prints. 2018;1802.03426. ↩︎
- Satomi et al. Pathol Int. 2023 73(10):523-526. ↩︎
- Shibayama T et al. BMC Pulm Med. 2023;23(1):324. ↩︎
- 里見介史. WHO2021におけるDNAメチローム解析. Clinical Neuroscience. 2023;41(4):463-7. ↩︎
- 竹内賢吾. 循環プロセスとしての病理学. 病理と臨床. 2017;35(12):1170-1. ↩︎
- Samuel Eilenberg SM. General Theory of Natural Equivalences. Transactions of the American Mathematical Society. 1945;58(2):231-94. ↩︎
- Satomi et al. Neuro Oncol. 2022;24(5):834-46. ↩︎
- Nagane M et al. Cancers (Basel). 2022;14(22). ↩︎
- Satomi K, Ichimura K, Shibahara J. Pathol Int. 2024 74(2):51-67. ↩︎

